Pourquoi faut-il identifier les nouveaux variants ?
Alors que le Pr Delfraissy parle d’échappement par pression de sélection suite à une immunité collective naturelle avec l’exemple de Manaus et du Cap, se pose la question d’un échappement des nouveaux variants du SARS-CoV-2 à la réponse immunitaire en plus de leur plus grande transmissibilité. Un des avantages des vaccins ARN est qu’il est facile de remplacer l’ARN des souches plus anciennes de virus par de nouvelles séquences, voire plusieurs séquences, correspondant aux nouveaux variants. Leur surveillance est donc essentielle.
En pratique, que doivent faire les laboratoires pour les identifier ?
Depuis le DGS urgent du 23 janvier, les laboratoires de biologie médicale doivent conserver les prélèvements ayant donné lieu à une RT-PCR afin de permettre la réalisation d’un second test de criblage aux variantes, et sont tenus d’envoyer sans délai aux plateformes réalisant ce deuxième test les prélèvements ayant donné lieu à un résultat de RT-PCR positif. En effet, des kits RT-PCR spécifiques des variantes sont utilisables à partir du samedi 23 janvier 2021 sur l’ensemble du territoire (la liste est intégrée sur la plateforme du MSS et sera mise à jour régulièrement ) pour réaliser des tests RT-PCR de criblage de variantes, sur la base d’un premier test RT-PCR positif.
Quand doit on réaliser un séquençage ?
S’agissant des séquençages, les prélèvements doivent être orientés de la manière suivante :
-Les prélèvements réalisés sur des personnes de retour de l’étranger sont séquencés en priorité, uniquement s’ils n’ont pu faire l’objet d’un deuxième test RT-PCR réalisé avec un kit criblant les variantes, dans le réseau des laboratoires de biologie médicale disposant de la capacité de séquençage ou d’autres capacités de séquençage identifiées par les ARS et répondant au cahier des charges du CNR.
-Les prélèvements issus des personnes contaminées en clusters ou dans des zones présentant une augmentation forte et inexpliquée du TI sont orientés vers les laboratoires liés au réseau CNR-ANRS. Dans ces situations, l’envoi de 5 à 6 prélèvements avec un Ct <28 est nécessaire et suffisant, accompagné des renseignements clinico-épidémiologiques utiles tel que recommandé par le CNR.
-Des enquêtes Flash aléatoires, seront répétées à intervalle très régulier, avec pour objectif d’enrichir une base nationale de séquences permettant des analyses régulières à la recherche de nouveaux variants.
Quelle est la situation actuelle ?
Point au 18 mars : Augmentation de la proportion de suspicions de variants d’intérêt 20I/501Y.V1 à 72% (supérieures à 50% dans 91 départements métropolitains), stabilisation des suspicions de variant 20H/501Y.V2 ou 20J/501Y.V3 à 5%
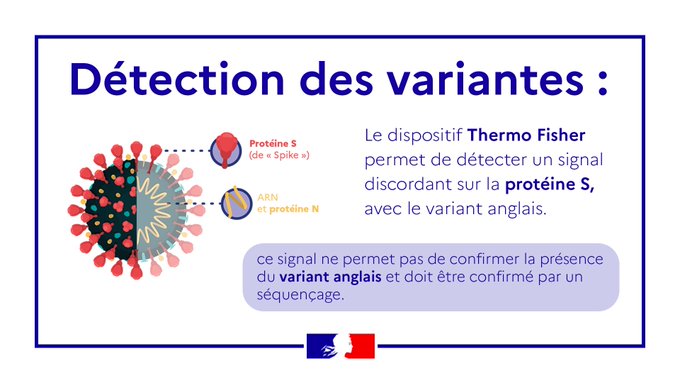
Pourquoi ne doit-on plus utiliser Thermofisher comme technique de criblage
Jusqu’à maintenant le seul moyen de détecter les variantes sur les prélèvements positifs était de réaliser une deuxième PCR avec un dispositif particulier (Thermo Fisher).
Ce dispositif est très utile mais manque de spécificité et la suspicion de variants devait être confirmée par un séquençage. Ce réactif peut continuer à être utilisé en PCR de 1er intention mais la PCR de criblage doit être réalisée avec un réactif spécifique. La liste des réactifs est disponible sur le site du ministère.